曼哈顿图(manhattan plot),顾名思义,是一种类似曼哈顿摩天大楼排列的一种展示图。不知道曼哈顿长什么样?我们来看看:

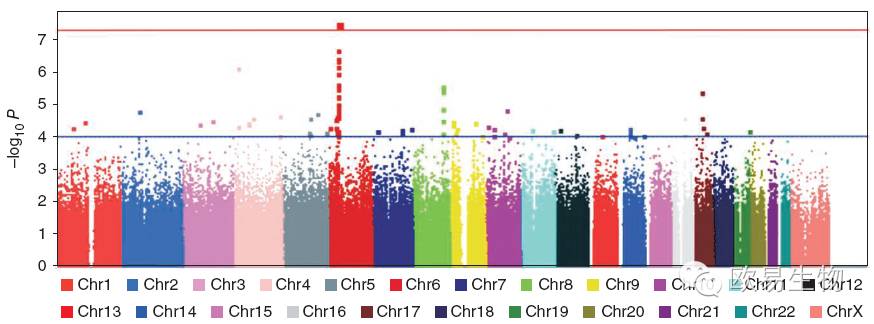
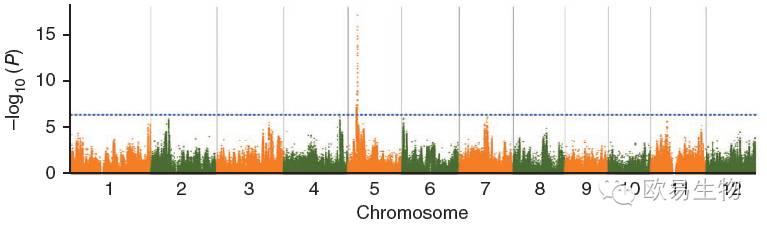
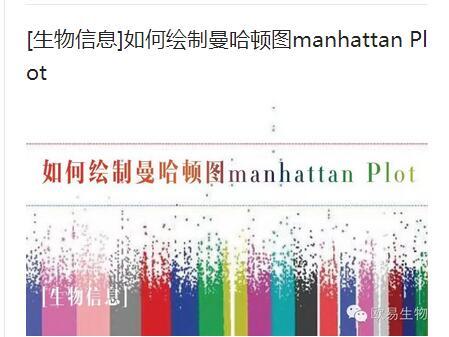
在生物和统计学上,做频率统计、突变分布、GWAS关联分析的时候,我们经常会看到一些非常漂亮的manhattan plot,能够对候选位点的分布和数值一目了然,如下:
说到绘图,虽然manhattan plot属于比较简单的散点图了,但想要达到发文章的级别,用excel一点一点去画就不是太合适啦。在这里我们推荐用强大的R语言,但是很多人想到代码和命令行就头疼,即使是网上有现成的R脚本也是,怎么办呢?

这次就给大家推荐一款简单实用的软件,这个软件并不陌生,如果是做关联分析应该都听说过:Haploview,对,就这个做连锁图的软件,java写的小程序,windows下就能运行,非常轻便。
下载地址:
https://www.broadinstitute.org/ftp/pub/mpg/haploview/Haploview.jar
Haploview的详细用法很多,我们这里只关注如何画manhattan plot吧,首先双击运行haploview.jar,打开如下的主界面(如果主界面默认是空白,就用file里面的open new data)。
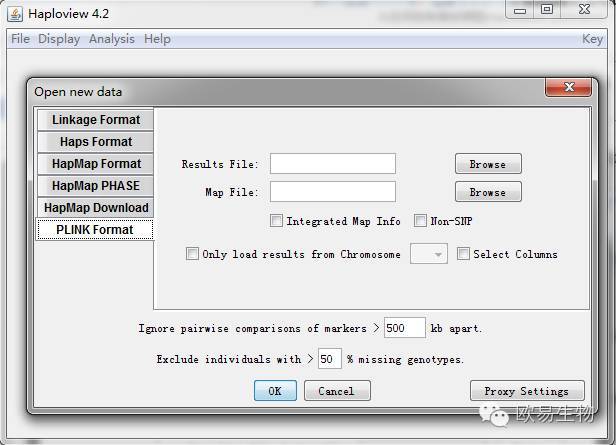
接下来我们把之前的数据导入,haploview支持多种数据格式,如果是关联分析相关的,推荐使用plink format。如果恰好你是用plink做的关联分析,那么恭喜,直接将对应的assoc文件和map文件导入上面的两个框就可以了。但……还是假设我们没有……假设我们手上只有位点坐标和pvalue(或者频率、比例什么的),那么我们需要伪造这两个文件了。

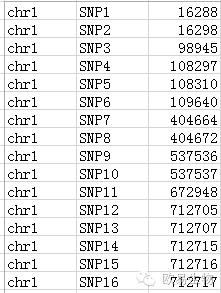
首先,map文件至少包含三列——染色体号,SNP名字,SNP物理位置。如下图所示,我们把这三列做好,然后存为纯文本格式,随便命名为fake.map,因为是假的嘛……
其次,assoc文件,虽然plink直接跑出来的会有一大堆,但是,实际只要两列就够了……SNP名字和pvalue,区别是顶上要有标题行,如下图所示,我们同样做好这两列,然后命名为fake.assoc。注意如果一个数值缺失了,可以写成NA。
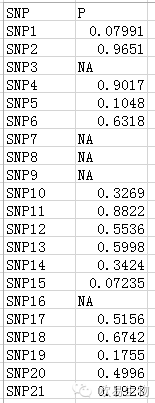
文件做好了,我们在haploview里选plink format,然后导入这两个文件,点OK,就会出现一个下面的SNP列表:
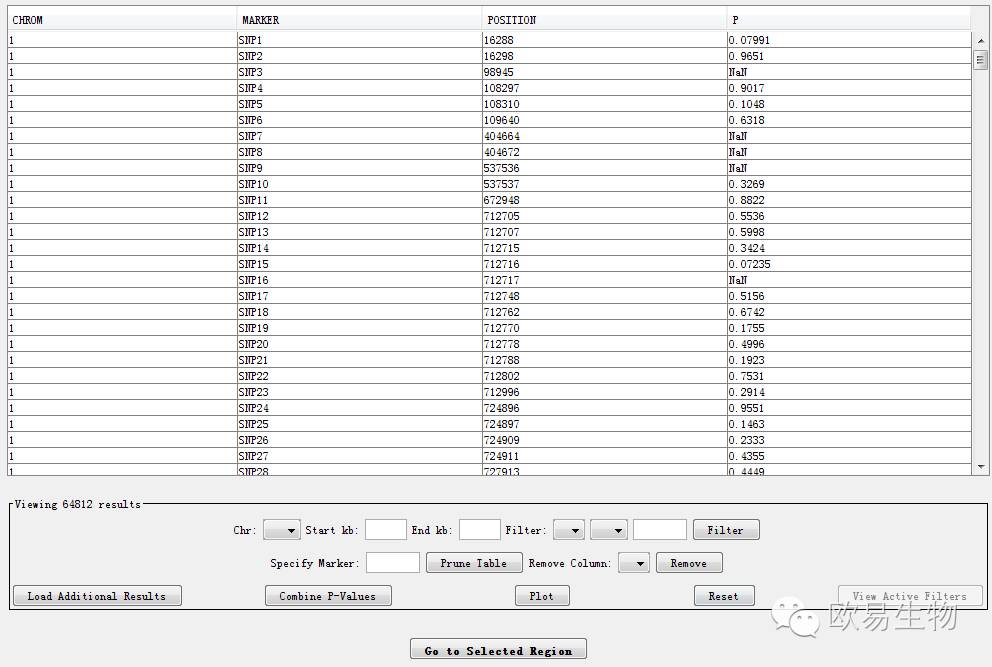
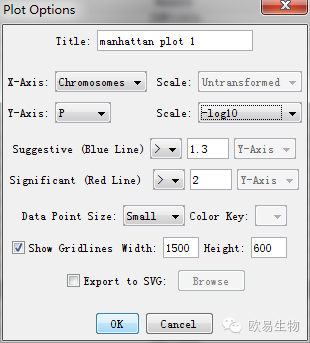
其他不多解释了,我们简单粗暴直接点Plot按钮,进入参数选择,这里的选项都很实用:第一行是图片标题,然后让你选择X,Y轴分别用哪一列来画,X轴显然用染色体,Y轴我们选择pvalue那一列,也就是P,尺度我们使用常用的-log值,否则出来的图会很杂乱。
再下面两行表示:是否添加表示显著性的标线,一条蓝色,一条红色,同时,超出这个标线的点会进行加粗处理,这里可根据实际需要调整,不需要就不用填。
为了演示起见,我们假定pvalue<0.05为比较显著,pvalue<0.01为非常显著,取-log之后对应的值分别是大于1.3和2。再下面是数据点的大小,点多的时候选Large要慎重。再下面的复选框表示是否添加栅格线。右边决定整个图形的宽度和高度,可自定义,如果染色体较多可以写长一些。最下面的选项是问是否导出到SVG格式,一般不需要。
以上,我们已经完成了所有准备和设置,点击OK看看自己的成果吧!喜欢吗?
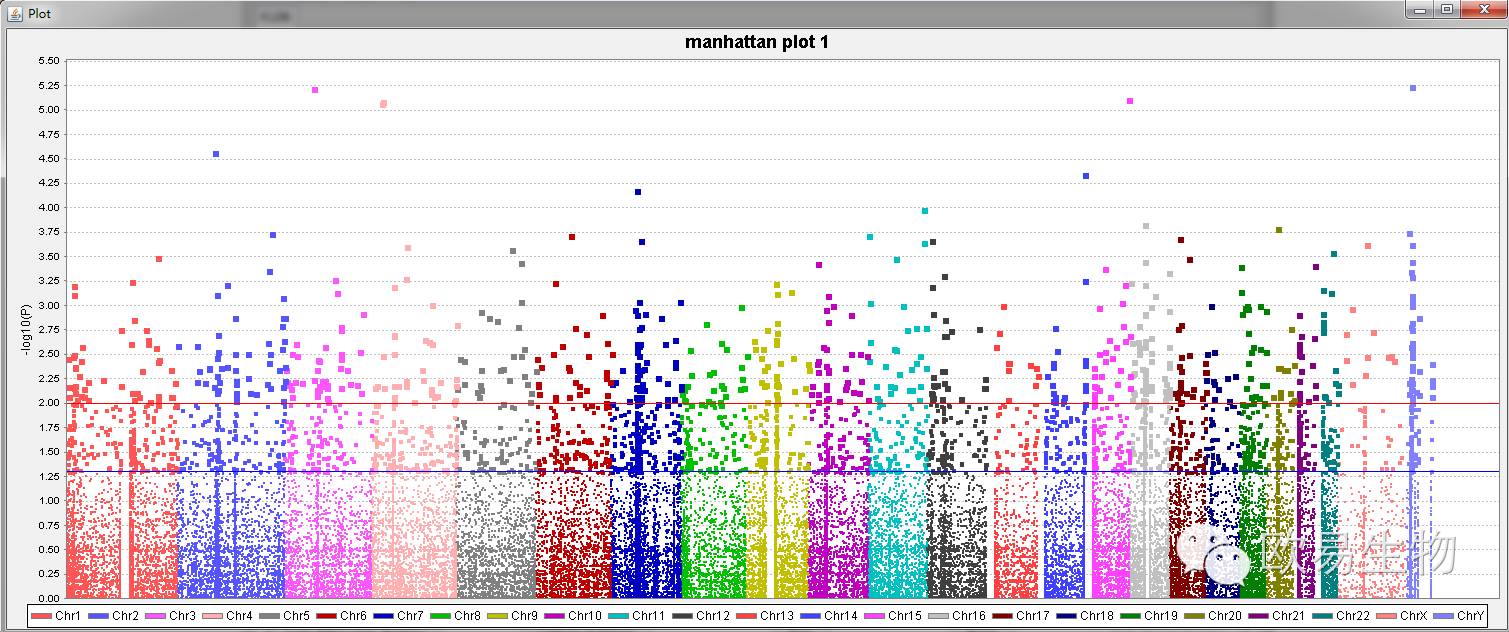
通过点击右键,我们还可以对图像进行一些细微调整,这些就有待大家去研究了。
 )
)






























我来说两句排行榜